よくある質問−DNAについて
よくある質問 − DNAについて
ニッポンジーンによくお問い合わせいただく内容を、Q&A集としてまとめております。
よくある質問
オリゴ合成
A. 実際の合成スケールと保証収量に関しては各合成サービスの製品仕様の表をご覧ください。
合成スケールは合成スタート時に使用する塩基のモル数です。したがって、そのすべての塩基の合成がうまくいった場合の収量は合成スケールで示した通りとなります。しかし、実際には合成機によって合成のカップリング効率が異なり、オリゴの鎖長が長ければ長いほど、合成途中でストップしたオリゴの混入する可能性が高くなりますので実際に得られるモル数とは異なっています。弊社ではさらに精製の回収率を考慮に入れて保証収量を設定しております。
50 mer以上のオリゴのカートリッジ精製については0.2 μmolスケールと1 μmolスケールになります。これは長鎖オリゴの合成効率、あるいは精製の回収率を考えると0.05μmolスケールでは十分な収量を得られない可能性があるからです。
A. ニッポンジーンでは、定量、質量分析装置によって分子量、完全長のオリゴと副産物について測定しています。また、Double-Dye Probeについては分析用HPLCによるQCも行っています。
全てのオリゴヌクレオチドの収量を測定した後、収量不足、または質量分析結果より私どもの基準に合わないと判定されたオリゴは即日再合成となります。それらの結果はお客様へ高品質なオリゴをお届けするためばかりでなく、今後も続けてそのような高品質の製品をお届けするための貴重なデータともなります。このような品質管理/フィードバックによって更なる品質の向上を目指しております。
A. 合成機への配列の入力はLANを用いてオーダー組み込み時に行われます。LANは物理的に外部のネットワークから遮断されておりますので機密性の高い配列でも安心です。合成後に疑問が生じましても合成機の合成ログによって合成機がカップリングした塩基を確認することができます。
A. ニッポンジーンでは、ほとんどのオリゴヌクレオチドを100 μM水溶液品もしくは乾燥品として室温で発送しております。室温での輸送は品質に影響を与えませんが、お手元にお届き次第、凍結保存をしてください。
水溶液品については、凍結融解の繰り返しは絶対に避けるべきものと思われます。実験に必要な容量分を小分けにし、-20℃にて保存することをお勧めします。
A. 私どもの合成機のカップリング効率で理論的に計算しますと100 merから200 merまでの合成が精製の際の収率を考えても可能のように思えます。
しかしながらたとえ私どもで200 merを合成し、収率よく精製を行ったとしても200 mer合成には問題があるといわざるを得ません。合成サイクルのステップごとに塩基にダメージが加えられその数は塩基のカップル数に応じて増えていきます。この場合200回のサイクル分だけその確率が高くなるとお考えください。このような塩基への修飾・ダメージは変異を起こし、誤った塩基をコードする製品となります。この製品を取り除くような精製は一般には困難ですので、お客様のほうで長いオリゴを注文される場合はそれを考慮されてご注文いただければ幸いです。
現実的には200 merを合成するのではなく、100 merを2本合成、短い相補鎖を用いて2本のオリゴのライゲーションを行い200 merを作成する方法が考えられます。必要に応じてこの長鎖にリン酸基付加を行うこともできます。
A. 次の式を用いてオリゴヌクレオチドの分子量を計算いたします。
(nA x 313.2109) + (nG x 329.2103) + (nC x 289.1858) + (nT x 304.19757) + (nD x 315.53959) + (nM x 301.195) + (nH x 302.19809) + (nW x 308.70424) + (nR x 321.2106) + (nY x 296.69169) + (nV x 310.53567) +(nS x 309.19805) + (nK x 316.70394) + (nN x 308.9511425) + (nB x 307.53122) – (61.96459)
ただし、上記計算式中のコードは以下を示します。 nD=A/G/T, nM=A/C, nH=A/C/T, nW=A/T, nR=A/G, nY=C/T, nV=A/C/G, nS=C/G, nK=G/T, nN=A/G/C/T, nB=C/G/T
A. ここでのTm値は、修飾を持たない通常のホスホジエステル結合のDNA用に計算されています。この計算値は、ビオチン、ジゴキシゲニン、蛍光色素、アミンといった修飾ヌクレオチドは考慮しておりませんが、実験条件を決める際の参考としてご参照ください。
Tm値はオリゴ水溶液のカチオン濃度やホルムアミドによって融解温度が左右されるため、特にTmの計算にはNa+カチオンを考慮に入れた次の式を用いております。
Tm=81.5 + 16.6log[Na+] (= – 21.597098) + 0.41(%G + %C) -675/length
(Baldino et al. Methods in Enzymology. (1989), 168, 761-777).
弊社では[Na+]=50 mMとしてTm値を計算しております。お客様のほうで異なる計算式を用いる場合はその結果と合わないことがございます。実際に実験を行う時のリファレンスとして保管ください。
A. G-richなオリゴに関しては特別な合成法が必要となりますが、合成自体には問題はありません。その際、カートリッジ精製を行うことをお勧めいたします。
G-richなオリゴを取り扱う場合は同様に使用法にもご注意ください。G-richなオリゴは凝集し、テトラマー構造をとりやすく、このことが水溶液中で沈殿を生じる原因となります。これを避けるためにはバッファーに溶解させて相補するターゲット鎖を加えた状態で95℃まで温度を上げ、ゆっくりと温度を下げていく方法をとることもひとつといえるでしょう。
A. 私どもがお届けしているオリゴヌクレオチドのうち、大きなスケールでお届けする時には凍結乾燥を行い、またODを測定して収量をお知らせしています。私どもがmgで秤量しない理由として、DNAの凍結乾燥サンプルは水分をDNA duplex内のmajor groove, minor grooveに挟み込む形や塩基・糖・リン酸基への水和物として含んでいます。したがって、凍結乾燥品は実際には50%もの水分をその重量の中に含んでいるのです。
A. はい、PCRに適しています。
A. Taq DNA polymeraseを用いたPCR産物は0.25%のエラーを起こすことが知られており、また組換えベクター/宿主系の中で「self correcting」エラーが出るケースもあります。理論的には長い塩基を合成した場合にも合成サイクルの増加に伴ってプリン基などのダメージを受けることも考えられますが、Taq DNA Polymeraseを使われていないのにそのようなトラブルが起きました場合、直ちにお申し付けいただければ無料で再合成を行います。
A. 等モル混合塩基であれば3’末端、配列中を含めて可能です。
A. 0.2 μmoleスケールでの合成効率は0.05 μmoleスケールの場合と必ずしも同じとは限らず、スタート時のモル数が4倍であっても収量も4倍になるとは限りません。
精 製
A. 脱塩 70%、Cartridge 75%、HPLC 85%、PAGE 90% です。これは目安であり保証ではございません。
A. 精製は大まかに分けて脱塩、カートリッジ精製、HPLC精製を用意しております。また長鎖オリゴにはPAGE精製が適用できます。どの程度の精製が必要かは実験の目的によって大きく異なることが多いですが、簡単にまとめると下のようになります。
| 用途 | 精製 |
|---|---|
| DNA増幅(PCR) | 脱塩 |
| シークエンシング | 脱塩、カートリッジ、HPLC |
| ハイブリダイゼイションプローブ | 35 mer以上は精製(カートリッジまたはHPLC)、34 mer以下は脱塩、もしくは精製(カートリッジまたはHPLC) |
| 変異導入 | 精製(カートリッジまたはHPLC) |
| 遺伝子合成 | 精製(カートリッジまたはHPLC) |
| DNA構造解析 | 精製(カートリッジまたはHPLC) |
| リンカー、アダプター構築 | 精製(カートリッジまたはHPLC) |
A. HPLC精製は精製度についての詳細なレポートを記録することができるため、合成・精製過程における問題点も発見することができます。また、修飾オリゴの中にはカートリッジで精製することのできないものもあります(HEX-labelされたオリゴヌクレオチドなど)。
A. ニッポンジーンのオリゴは無料で脱塩されております。短いオリゴにはこの精製法が充分な精製度ですが、次のような場合にはHPLC精製が特に必要とされています。
- 化学修飾されたオリゴ : HPLC精製は修飾されていないオリゴを取り除くことができます。DIG標識、ビオチン標識、蛍光標識の場合がこれに当てはまります。
- 長鎖オリゴ : 合成は高いTurnoverで行われておりますので常に鮮度の高い試薬が用いられており、弊社の合成機を用いた場合、固相DNA合成のカップリング効率は99%以上です。この場合完全長のcrude 50 merの収率は60%強、100 merの場合約40%になります。これらの収率を同じカップリング効率で合成されたcrude 20 mer(約83%)と比較すると短鎖オリゴは相対的に純度が高いことになります。このことより、カートリッジないしはHPLCによる精製は、短鎖の場合にはお使いになられなくてもPCRグレードには充分ですが、長鎖オリゴ(49 – 99 mer)をご利用の際は精製が必要となります。長鎖オリゴ同様の理由で、弊社では35 mer以上(収率約70%)のオリゴにはカートリッジ精製をご利用にいただきますようお勧めいたしております。
- アンチセンスDNA : アンチセンスDNAに不純物が混入しますと組織細胞の成育の阻害をまねき不正確な実験結果につながることがあります。アンチセンスDNAにはHPLC精製を行うのが良い選択肢と思われます。
- RNA : ニッポンジーンのRNA合成は高い品質を誇りますが脱保護などの工程で不純物のトレースが残る場合があります。HPLCによるRNAの精製は高い力価を得るためには必須であるといえます。
修飾オリゴ
A. はい、スタンダードオリゴはフリーの5’ -、3’ -水酸基を持ちますので5’ -キナーゼによるラベル、あるいはヌクレオシド3-リン酸とターミナルトランスフェラーゼを使った3’ -ラベリングも可能です。
A. はい、できます。
A. 原材料が高価なことがまず一点、それに加えて修飾試薬の安定性が低く、長時間の保存が困難なことが最大の原因です。
A. 弊社の5’ Biotin-TEG修飾は、Biotin-TEGホスホアミダイトを用いております。TEGは、通常のBiotinホスホアミダイトのリンカーより長く、また、内部に酸素原子を有していることから立体構造的な自由度が高いという特長をもっています。この特長によって、 BiotinがStreptavidinと結合する際の立体障害が低減し、Biotin-Streptavidinの結合効率が上がることが知られています。
A. 核酸医薬として注目を集めているアンチセンス核酸とは何か、siRNAとの違いについて解説された、ニッポンジーンマテリアル社のホームページに掲載の記事がご参考になるかと思います。
「アンチセンス核酸 vs. siRNA」(ニッポンジーンマテリアル社ホームページ)
A. 核酸医薬の一種として近年注目を集めているアンチセンス核酸の構造と合成について解説された、ニッポンジーンマテリアル社のホームページに掲載の記事がご参考になるかと思います。
「アンチセンス核酸の合成」(ニッポンジーンマテリアル社ホームページ)
Double-Dye Probe
A. プローブがしばらくの間、光などにさらされ色素が脱離してしまった可能性があります。この場合PCRの反応自体は進んでも蛍光色素がありませんのでデータを得ることはできません。蛍光色素などで修飾されたオリゴヌクレオチドは-20℃、冷暗所にて保存ください。凍結・融解の繰り返しは避けてください。
A. DNaseなどのコンタミネーションが起こりますとDNAの分解に伴って蛍光色素の脱離が起こり、高いバックグラウンドにつながります。オリゴヌクレオチドの分注の際には滅菌されたチップとチューブを使われてDNaseなどの混入を極力避けてご利用ください。
もしくは、Eclipse®Dark Quencherをお勧めします。Eclipse®Dark Quencherは蛍光を発しないため測定時のバックグラウンドが低くなり、より精度の高い実験が可能となります。
A. Yakima-Yellow®の最大蛍光波長(549 nm)、励起波長(530.5 nm)はVIC®の最大蛍光波長、励起波長ともに非常に近いためVIC®用のチャンネルとフィルターのセッティングをそのまま利用することができます。
Yakima Yellow® – Eclipse® Dark Quencherは VIC® – TAMRAに比べて増幅曲線がシャープで、対数増幅期が長い。
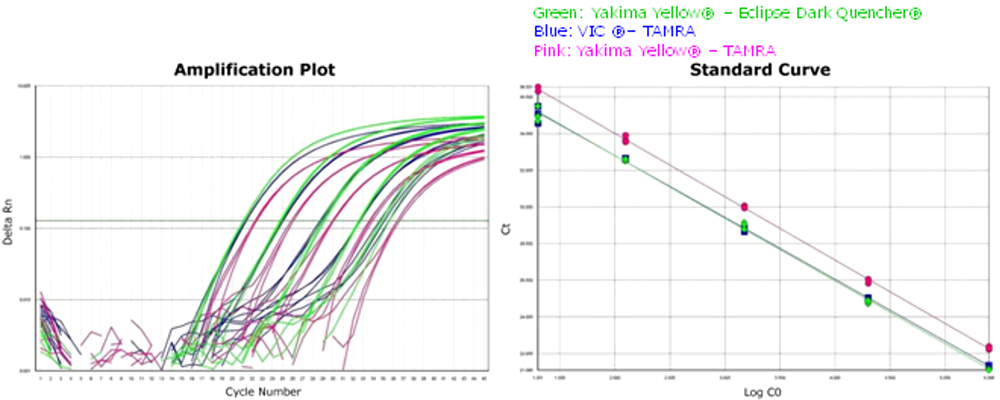
サーマルサイクラー:ABI 7500 Fast System: 9600 emulation Mode
テンプレートDNA: GMトウモロコシプラスミドセット
プライマー:トウモロコシ内在性DNA SSIIb-3オリゴヌクレオチド
ダブルダイプローブ:Yakima Yellow® – Eclipse® Dark Quencher, VIC® – TAMRA, Yakima Yellow® – TAMRA
A. 一反応で複数の変異を検出する際にマルチプレックス反応は有用です。現在2種のターゲットを検出するのにもっとも頻繁に用いられている組み合わせがFAMとHEX(及びJOE、一般に高価)、Passive ReferenceとしてROXです。しかしながらFAM/HEXとFAM/JOEは感度が多少低いことが知られています。
当社ではVICやJOEに代わる色素としてYakima Yellowを取り扱っております。Yakima Yellowは蛍光強度、分光分解能の面で優れており、蛍光強度の上昇によって、低いコピー数のampliconでも低いCt値(高い検出度)を得ることができます。
A. 6- Carboxytetramethylrhodamineです。




